研究チーム
Team研究チーム
研究項目A01 計画研究:ケモテクノロジーによるユビキチンコードの解読と制御
ケモテクノロジーの分子基盤を創出するユビキチン構造生物学
研究代表者 深井 周也京都大学大学院理学研究科化学専攻 教授 |
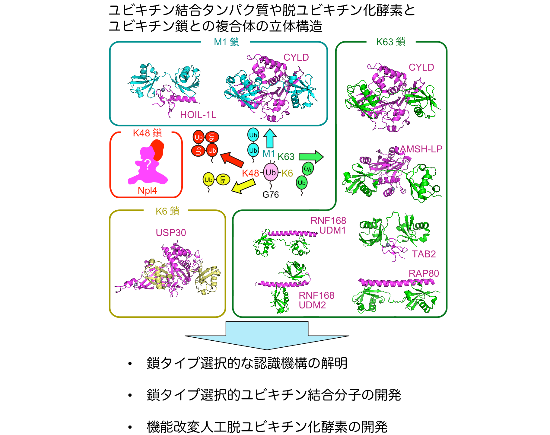
本計画研究の概要
ユビキチン修飾は、プロテアソームによるタンパク質分解のシグナルとしての役割にとどまらず、炎症シグナルやDNA修復などの多様な細胞機能の制御に必須な役割を担っています。ユビキチンは、数珠繋ぎになったユビキチン鎖として機能する例が多く知られていますが、繋がり方の多様性にリン酸化などの別の翻訳後修飾も加わって、ユビキチン修飾が持つ機能情報は想定を超えた複雑さを呈してきています。本研究では、ユビキチン結合タンパク質や脱ユビキチン化酵素とユビキチン鎖との複合体の立体構造を決定して、未解明のユビキチン鎖認識機構を原子レベルで明らかにします。また、本研究領域内で創出されるユビキチンシグナルを制御する機能性化合物の作用機構解明に必要な立体構造解析を行い、機能性化合物の高効率化・高機能化のための構造基盤を確立します。さらに、脱ユビキチン化酵素やユビキチン結合タンパク質を改変し、ユビキチン研究に有用な分子ツールの開発を目指します。
1. 立体構造解析による分子基盤の創出
特定のユビキチン鎖に選択性のある既知の脱ユビキチン化酵素やユビキチン結合ドメイン、さらに領域内で見出される新規ユビキチン結合ドメインとユビキチン鎖との複合体やユビキチンが介する制御因子などの構造解析を通じて、鎖タイプ識別の新たな分子機構を解明し、他の計画研究に情報提供します。また、本領域研究で創出される機能性化合物との共結晶構造解析により機能性化合物の高効率化・高機能化のための構造基盤を確立します。
2. 鎖タイプ選択的ユビキチン結合分子の開発
K63鎖に選択的に結合するRAP80のタンデムUIMやRNF-168のUDM1やUDM2などのα-ヘリックスを骨格とする構造情報を元に、ステープル化技術も活用して、鎖タイプ選択的なユビキチン鎖結合分子を設計・開発します。
3. 機能改変人工脱ユビキチン化酵素の開発
プロテアソームの脱ユビキチン化活性を担うRpn11-Rpn8複合体は、ユビキチン鎖をen blocにタンパク質から切断します。Rpn11-Rpn8複合体を改変することで、多様なユビキチン化基質からユビキチン鎖を切り出す人工酵素を創出し、ユビキチン鎖高次構造解析に有用な分子ツールを開発します。
研究概要を示す模式図
本領域での研究成果
- Okatsu K, Sato Y, Yamano K, Matsuda N, Negishi L, Takahashi A, Yamagata A, Goto-Ito S, Mishima M, Ito Y, Oka T, Tanaka K, *Fukai S.
Structural insights into ubiquitin phosphorylation by PINK1.
Sci. Rep. 8, 10382 (2018)
PMID: 29991771 - Takahashi TS, Sato Y, Yamagata A, Goto-Ito S, Saijo M, *Fukai S.
Structural basis of ubiquitin recognition by the winged-helix domain of Cockayne syndrome group B protein.
Nucleic Acids Res. 47, 3784-3794 (2019)
PMID: 30753618 - Sato Y, Tsuchiya H, Yamagata A, Okatsu K, Tanaka K, *Saeki Y, *Fukai S.
Structural insights into ubiquitin recognition and Ufd1 interaction of Npl4.
Nat. Commun. 10, 5708 (2019)
PMID: 31836717
プレスリリース
http://www.iam.u-tokyo.ac.jp/press_release/191213/
http://www.igakuken.or.jp/topics/2019/1213.html
http://www.igakuken.or.jp/english/topics/2019/e-1213.html - Yamanaka S, Sato Y, Oikawa D, Goto E, Fukai S, Tokunaga F, *Takahashi H, *Sawasaki T.
Subquinocin, a small molecule inhibitor of CYLD and USP-family deubiquitinating enzymes, promotes NF-κB signaling.
Biochem. Biophys. Res. Commun. 524, 1-7 (2020)
PMID: 31898971 - Oikawa D, Sato Y, Ohtake F, Komakura K, Hanada K, Sugawara K, Terawaki S, Mizukami Y, Phuong HT, Iio K, Obika S, Fukushi M, Irie T, Tsuruta D, Sakamoto S, Tanaka K, Saeki Y, Fukai S, *Tokunaga F.
Molecular bases for HOIPINs-mediated inhibition of LUBAC and innate immune responses.
Commun. Biol. 3, 163 (2020)
PMID: 32246052 - *Takahashi H, Yamanaka S, Kuwada S, Higaki K, Kido K, Sato Y, Fukai S, Tokunaga F, *Sawasaki T.
A Human DUB Protein Array for Clarification of Linkage Specificity of Polyubiquitin Chain and Application to Evaluation of Its Inhibitors.
Biomedicines 8, 152 (2020)
PMID: 32512835 - Li Y, Okatsu K, Fukai S, *Sato Y.
Structural basis for specific recognition of K6-linked polyubiquitin chains by the TAB2 NZF domain.
Biophys J. 120, 3355-3362 (2021)
PMID: 34242591
プレスリリース
関連する代表的な論文
- Sato Y, Yoshikawa A, Yamagata A, Mimura H, Yamashita M, Ookata K, Nureki O, Iwai K, Komada M, *Fukai S.
Structural basis for specific cleavage of Lys 63-linked polyubiquitin chains.
Nature 358-362 (2008)
PMID: 18758443 - Sato Y, Yoshikawa A, Mimura H, Yamashita M, Yamagata A, *Fukai S.
Structural basis for specific recognition of Lys 63-linked polyubiquitin chains by tandem UIMs of RAP80.
EMBO J. 28, 2461-2468 (2009)
PMID: 19536136 - Yoshikawa A, Sato Y, Yamashita M, Mimura H, Yamagata A, *Fukai S.
Crystal structure of the NEMO ubiquitin-binding domain in complex with Lys 63-linked di-ubiquitin.
FEBS Lett. 583, 3317-3322 (2009)
PMID: 19766637 - Sato Y, Yoshikawa A, Yamashita M, Yamagata A, *Fukai S.
Structural basis for specific recognition of Lys 63-linked polyubiquitin chains by NZF domains of TAB2 and TAB3.
EMBO J. 28, 3903-3909 (2009)
PMID: 19927120 - Sato Y, Fujita H, Yoshikawa A, Yamashita M, Yamagata A, Kaiser SE, *Iwai K, *Fukai S.
Specific recognition of linear ubiquitin chains by the Npl4 zinc finger (NZF) domain of the HOIL-1L subunit of the linear ubiquitin chain assembly complex.
Proc. Natl. Acad. Sci. U.S.A. 108, 20520-20525 (2011)
PMID: 22139374 - Sato Y, Yamagata A, Goto-Ito S, Kubota K, Miyamoto R, *Nakada S, *Fukai S.
Molecular basis of Lys-63-linked polyubiquitination inhibition by the interaction between human deubiquitinating enzyme OTUB1 and ubiquitin-conjugating enzyme UBC13.
J. Biol. Chem. 287, 25860-25868 (2012)
PMID: 22679021 - Sato Y, Goto E, Shibata Y, Kubota Y, Yamagata A, Goto-Ito S, Kubota K, Inoue J, Takekawa M, Tokunaga F, *Fukai S.
Structures of CYLD USP with Met1- or Lys63-linked diubiquitin reveal mechanisms for dual specificity.
Nat. Struct. Mol. Biol. 22, 222-229 (2015)
PMID: 25686088 - Sato Y, Okatsu K, Saeki Y, Yamano K, Matsuda N, Kaiho A, Yamagata A, Goto-Ito S, Ishikawa M, Hashimoto Y, Tanaka K, *Fukai S.
Structural basis for specific cleavage of Lys6-linked polyubiquitin chains by USP30.
Nat. Struct. Mol. Biol. 24, 911-919 (2017)
PMID: 28945247 - Takahashi TS, Hirade Y, Toma A, Sato Y, Yamagata A, Goto-Ito S, Tomita A. *Nakada S, *Fukai S.
Structural insights into two distinct binding modules for Lys63-linked polyubiquitin chains in RNF168.
Nat. Commun. 9, 170 (2018)
PMID: 29330428





