研究チーム
Team研究チーム
研究項目A02 計画研究:革新的ユビキチンケモテクノロジーの創出
ユビキチン機能制御のためのケミカルバイオロジー
研究代表者 吉田 稔国立研究開発法人理化学研究所環境資源科学研究センター ケミカルゲノミクス研究グループ グループディレクター |
|
研究分担者 川谷 誠国立研究開発法人理化学研究所環境資源科学研究センター 生命分子解析ユニット 専任研究員 |
|
研究分担者 近藤 恭光(2021年度まで)国立研究開発法人理化学研究所環境資源科学研究センター ケミカルバイオロジー研究グループ 専任研究員 |
|
研究分担者 Pradipta Ambara東京工業大学物質理工学院応用化学系 助教 |
本計画研究の概要
タンパク質ユビキチン化は、プロテアソームによるタンパク質分解のみならず、当初の想定を大きく超える多様なタンパク質機能制御機構であることがわかってきました。この多様な機能を生み出す基盤は、ユビキチン修飾の構造多様性と相互作用する分子(デコーダー)の多様性にあります。その複雑性ゆえに全体像を理解するためには、従来の方法では限界がありました。本新学術領域研究では、化学的アプローチ(ケモテクノロジー)を駆使して、ユビキチン修飾系を理解し、操作する新たな方法論を開拓しようとしており、本研究はその基盤となるツール開発を目的とするものです。
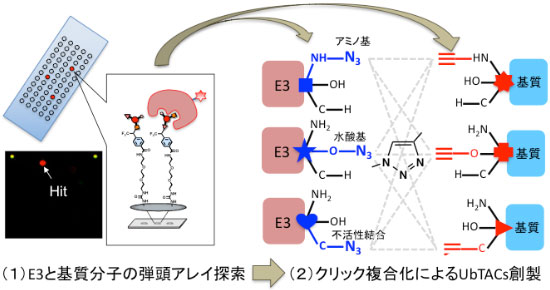
その中心となる技術は、UbTACsと名付けた新たなユビキチン化誘導キメラ分子です。SNIPERあるいはPROTACsと呼ばれるキメラ化合物によりユビキチンリガーゼをハイジャックすることで本来の標的分子以外の基質タンパク質(ネオ基質)を分解誘導する技術を発展させ、ユビキチンやデコーダー分子の特定の機能を瞬時に喪失させることが可能な分子プローブを創出し、ユビキチンの機能解析のためのツールを開発します。さらに、単にネオ基質のノックダウンだけでなく、エンドサイトーシス、シグナル伝達、オートファジーなどを誘導できる新しいユビキチン化誘導キメラ分子の創出を目指します。もう一つの重要なアプローチはユビキチン鎖とそれを認識するデコーダー分子との間の相互作用を阻害する分子プローブの発見です。これらを実現するために、われわれは、迅速な多検体スクリーニングを可能とする化合物アレイとE3リガンドとリガンド化合物をドッキングさせるための化合物官能基化技術、およびタンパク質間相互作用の高速スクリーニング系を確立しており、これを用いて複数のタンパク質を標的としたUbTACsと阻害剤開発を実現します。
研究概要を示す模式図
本領域での研究成果
- Sohtome Y, Shimazu T, Barjau J, Fujishiro S, Akakabe M, Terayama N, Dodo K, Ito A, Yoshida M, Shinkai Y, Sodeoka M.
Unveiling epidithiodiketopiperazine as a non-histone arginine methyltransferase inhibitor by chemical protein methylome analyses.
Chem. Commun. 54, 9202-9205 (2018)
PMID: 30063235 - Hiranaka S, Tega Y, Higuchi K, Kurosawa T, Deguchi Y, Arata M, Ito A, Yoshida M, Nagaoka Y, Sumiyoshi T.
Design, Synthesis, and Blood-Brain Barrier Transport Study of Pyrilamine Derivatives as Histone Deacetylase Inhibitors.
ACS Med. Chem. Lett. 9, 884-888 (2018)
PMID: 30258535 - Furuta H, Yoshihara H, Fukushima T, Yoneyama Y, Ito A, Worrall C, Girnita A, Girnita L, Yoshida M, Asano T, Komada M, Kataoka N, Chida K, Hakuno F, Takahashi SI.
IRS-2 deubiquitination by USP9X maintains anchorage-independent cell growth via Erk1/2 activation in prostate carcinoma cell line.
Oncotarget 9, 33871-33883 (2018)
PMID: 30338032 - Nakamoto Y, Pradipta AR, Mukai H, Zouda M, Watanabe Y, Kurbangalieva A, Ahmadi P, Manabe Y, Fukase K, Tanaka K.
Expanding the applicability of the metal labeling of biomolecules by the RIKEN click reaction: A case study with Gallium-68 positron emission tomography.
ChemBioChem 19, 2055-2060 (2018)
PMID: 30066425 - Simpkins SW, Nelson J, Deshpande R, Li SC, Piotrowski JS, Wilson EH, Gebre AA, Safizadeh H, Okamoto R, Yoshimura M, Costanzo M, Yashiroda Y, Ohya Y, Osada H, Yoshida M, Boone C, Myers CL.
Predicting bioprocess targets of chemical compounds through integration of chemical-genetic and genetic interactions.
PLoS Comput. Biol. 14, e1006532 (2018)
PMID: 30376562 - Tanei T, Pradipta AR, Morimoto K, Fujii M, Arata M, Ito A, Yoshida M, Saigitbatalova E, Kurbangalieva A, Ikeda JI, Morii E, Noguchi S, Tanaka K.
Cascade reaction in human live tissue allows clinically applicable diagnosis of breast cancer morphology.
Adv. Sci. 6, 1801479 (2018)
PMID: 30693189
プレスリリース - Mizutani A, Yashiroda Y, Muramatsu Y, Yoshida H, Chikada T, Tsumura T, Okue M, Shirai F, Fukami T, Yoshida M, Seimiya H.
RK-287107, a potent and specific tankyrase inhibitor, blocks colorectal cancer cell growth in a preclinical model.
Cancer Sci. 109, 4003-4014 (2018)
PMID: 30238564 - Simpkins SW, Deshpande R, Nelson J, Li SC, Piotrowski JS, Ward HN, Yashiroda Y, Osada H, Yoshida M, Boone C, Myers CL.
Using BEAN-counter to quantify genetic interactions from multiplexed barcode sequencing experiments.
Nat. Protoc. 14, 415-440 (2019)
PMID: 30635653 - Suvarna K, Honda K, Muroi M, Kondoh Y, Osada H, *Watanabe N.
A small-molecule ligand of valosin-containing protein/p97 inhibits cancer cell-accelerated fibroblast migration.
J. Biol. Chem. 294, 2988-2996 (2019)
PMID: 30610116 - Ling F, Bradshaw E, Yoshida M.
Prevention of mitochondrial genomic instability in yeast by the mitochondrial recombinase Mhr1.
Sci. Rep. 9, 5433 (2019)
PMID: 30931958 - Shirai F, Tsumura T, Yashiroda Y, Yuki H, Niwa H, Sato S, Chikada T, Koda Y, Washizuka K, Yoshimoto N, Abe M, Onuki T, Mazaki Y, Hirama C, Fukami T, Watanabe H, Honma T, Umehara T, Shirouzu M, Okue M, Kano Y, Watanabe T, Kitamura K, Shitara E, Muramatsu Y, Yoshida H, Mizutani A, Seimiya H, Yoshida M, Koyama H.
Discovery of Novel Spiroindoline Derivatives as Selective Tankyrase Inhibitors.
J. Med. Chem. 62, 3407-3427 (2019)
PMID: 30883102 - Shu K, Iwamoto N, Honda K, Kondoh Y, Hirano H, Osada H, Ohno H, Fujii N, Oishi S.
Development of mirror-image screening systems for XIAP BIR3 domain inhibitors.
Bioconjug. Chem. 30, 1395-1404 (2019)
PMID: 30888797 - Qi Y, Zhao X, Chen J, Pradipta AR, Wei J, Ruan H, Zhou L, Hsung RP, Tanaka K.
In vitro and in vivo cancer cell apoptosis triggered by competitive binding of Cinchona alkaloids to the RING domain of TRAF6.
Biosci. Biotechnol. Biochem. 83, 1011-1026 (2019)
PMID: 31074699 - Pradipta AR, Fujii M, Tanei T, Morimoto K, Shimazu K, Noguchi S, *Tanaka K.
Tetramethylrhodamine is an essential scaffold of azide probe in detecting cellular acrolein.
Bioorg. Med. Chem. 27, 2228-2234 (2019)
PMID: 31023557 - Murashima A, Shinjo K, Katsushima K, Onuki T, Kondoh Y, Osada H, Kagaya N, Shin-Ya K, Kimura H, Yoshida M, Murakami S, Kondo Y.
Identification of a chemical modulator of EZH2-mediated silencing by cell-based high-throughput screening assay.
J. Biochem. 166, 41-50 (2019)
PMID: 30690451 - Mitsui E, Yoshida S, Shinoda Y, Matsumori Y, Tsujii H, Tsuchida M, Wada S, Hasegawa M, Ito A, Mino K, Onuki T, Yoshida M, Sasaki R, Mizukami T.
Identification of ryuvidine as a KDM5A inhibitor.
Sci. Rep. 9, 9952 (2019)
PMID: 31289306 - Takase S, Kurokawa R, Kondoh Y, Honda K, Suzuki T, Kawahara T, Ikeda H, Dohmae N, Osada H, Shin-Ya K, Kushiro T, *Yoshida M, *Matsumoto K.
Mechanism of action of prethioviridamide, an anticancer ribosomally synthesized and post-translationally modified peptide with a polythioamide structure.
ACS Chem. Biol. 14, 1819-1828 (2019)
PMID: 31365229 - Yoshida K, Kondoh Y, Iwahashi F, Nakano T, Honda K, Nagano E, Osada H.
Abscisic acid derivatives with different alkyl chain lengths activate distinct abscisic acid receptor subfamilies.
ACS Chem. Biol. 14, 1964-1971 (2019)
PMID: 31497942 - Lodochnikova OA, Chulakova DR, Gerasimova DP, Litvinov IA, Pradipta AR, *Tanaka K, *Kurbangalieva AR.
Stereochemical features of the crystallization of eight-membered 1,5-diazaheterocycles with chiral aminoindanol fragments at nitrogen atoms.
J. Struct. Chem. 61, 119-125 (2020) - Chulakova DR, *Pradipta AR, Lodochnikova OA, Kuznetsov DR, Bulygina KS, Smirnov IS, Usachev KS, Latypova LZ, Kurbangalieva AR, *Tanaka K.
Facile access to optically active 2,6-dialkyl-1,5-diazacyclooctanes.
Chem. Asian J. 14, 4048-4054 (2019)
PMID: 31381243 - Suvarna K, Honda K, Muroi M, Kondoh Y, Osada H, *Watanabe N.
Measurement of ATPase activity of valosin-containing protein/p97.
Bio-protocol 10, e3516 (2020) - Suvarna K, Honda K, Muroi M, Kondoh Y, *Watanabe N, *Osada H.
Identification of target protein for bio-active small molecule using photo-cross linked beads and MALDI-TOF mass spectrometry.
Bio-protocol 10, e3517 (2020) - *Pradipta AR, Tanei T, Morimoto K, Shimazu K, Noguchi S, *Tanaka K.
Emerging technologies for real-time intraoperative margin assessment in future breast-conserving surgery.
Adv. Sci. 7, 1901519 (2020)
PMID: 32382473 - Han P, Shichino Y, Schneider-Poetsch T, Mito M, Hashimoto S, Udagawa T, Kohno K, Yoshida M, Mishima Y, Inada T, *Iwasaki S.
Genome-wide survey of ribosome collision.
Cell Rep. 31,107610 (2020)
PMID: 32375038 - Jo T, *Nishikori M, Kogure Y, Arima H, Sasaki K, Sasaki Y, Nakagawa T, Iwai F, Momose S, Shiraishi A, Kiyonari H, Kagaya N, Onuki T, Shin-Ya K, Yoshida M, Kataoka K, Ogawa S, *Iwai K, Takaori-Kondo A.
LUBAC accelerates B-cell lymphomagenesis by conferring resistance to genotoxic stress on B cells.
Blood 136, 84-697 (2020)
PMID: 32325488 - Sako K, Futamura Y, Shimizu T, Matsui A, Hirano H, Kondoh Y, Muroi M, Aono H, Tanaka M, Honda K, Shimizu K, Kawatani M, Nakano T, Osada H, Noguchi K, *Seki M.
Inhibition of mitochondrial complex I by the novel compound FSL0260 enhances high salinity-stress tolerance in Arabidopsis thaliana.
Sci. Rep. 10, 8691 (2020)
PMID: 32457324 - *Pradipta AR, Chang TC, *Tanaka K.
Enantioselective synthesis of cyclic and linear diamines by iminocycloaddition.
Chirality 32, 1160-1168 (2020)
PMID: 32621328 - Ikeda H, Muroi M, Kondoh Y, Ishikawa S, Kakeya H, Osada H, *Imoto M.
Miclxin, a novel MIC60 inhibitor, induces apoptosis via mitochondrial stress in β-catenin mutant tumor cells.
ACS Chem. Biol. 15, 2195-2204 (2020)
PMID: 32584541 - Kawamura T, Futamura Y, Shang E, Muroi M, Janning P, Ueno M, Wilke J, Takeda S, Kondoh Y, Ziegler S, Watanabe N, Waldmann H, *Osada H.
Discovery of small-molecule modulator of heterotrimeric Gi-protein by integrated phenotypic profiling and chemical proteomics.
Biosci. Biotechnol. Biochem. 84, 2484-2490 (2020)
PMID: 32867616 - Radwan MO, Ciftci HI, Ali TFS, Koga R, Tateishi H, Nakata A, Ito A, Yoshida M, Fujita M, *Otsuka M.
Structure activity study of S-trityl-cysteamine dimethylaminopyridine derivatives as SIRT2 inhibitors: Improvement of SIRT2 binding and inhibition.
Bioorg. Med. Chem. Lett. 30,127458 (2020)
PMID: 32755678 - Kobayashi H, Nishimura H, Kudo N, Osada H, *Yoshida M.
A novel GSK3 inhibitor that promotes self-renewal in mouse embryonic stem cells.
Biosci. Biotechnol. Biochem. 84, 2113-2120 (2020)
PMID: 32640867 - Fujita K, Kondoh Y, Honda K, Haga Y, Osada H, Matsumura C, *Inui H.
Pesticide treatment reduces hydrophobic pollutant contamination in Cucurbita pepo through competitive binding to major latex-like proteins.
Environ. Pollut. 266, 115179 (2020).
PMID: 32717636 - Sophonnithiprasert T, Aruksakunwong O, Tashiro E, Kondoh Y, Muroi M, Osada H, Imoto M, Watanapokasin R.
Interaction between goniothalamin and peroxisomal multifunctional enzyme type 2 triggering endoplasmic reticulum stress.
Heliyon 6, e05200 (2020)
PMID: 33102840 - Smirnov I, Sibgatullina R, Urano S, Tahara T, Ahmadi P, Watanabe Y, Pradipta AR, Kurbangalieva A, *Tanaka K.
A Strategy for Tumor Targeting by Higher-Order Glycan Pattern Recognition: Synthesis and In Vitro and In Vivo Properties of Glycoalbumins Conjugated with Four Different N-Glycan Molecules.
Small 16, 2004831 (2020)
PMID: 33079456 - Muguruma K, Pradipta AR, Ode Y, Terashima K, Michiba H, Fujii M, *Tanaka K.
Disease-associated acrolein: A possible diagnostic and therapeutic substrate for in vivo synthetic chemistry.
Bioorg. Med. Chem. 28, 115831 (2020)
PMID: 33199202 - Kobayashi H, Hatakeyama H, Nishimura H, Yokota M, Suzuki S, Tomabechi Y, Shirouzu M, Osada H, Mimaki M, *Goto Y, *Yoshida M.
Chemical reversal of abnormalities in cells carrying mitochondrial DNA mutations.
Nat. Chem. Biol. 171,335-343 (2021)
PMID: 33168978
プレスリリース - Yoshimoto R, Chhipi-Shrestha JK, Schneider-Poetsch T, Furuno M, Maxwell Burroughs A, Noma S, Suzuki H, Hayashizaki Y, Mayeda A, Nakagawa S, Kaida D, Iwasaki S, *Yoshida M.
Spliceostatin A interaction with SF3B1 limits U1 snRNP availability and causes premature cleavage and polyadenylation.
Cell Chem. Biol. 28, 1356-1365 (2021)
PMID: 33784500
プレスリリース - *Pradipta A R, Ahmadi P, Terashima K, Muguruma K, Fujii M, Ichino T, Maeda S, *Tanaka K.
Targeted 1,3-dipolar cycloaddition with acrolein for cancer prodrug activation.
Chem. Sci. 12, 5438-5449 (2021)
プレスリリース - Pradipta A R, *Tanaka K.
Application of acrolein imines to organic synthesis, biofunctional studies, and clinical practice.
Chem. Rec. 21, 646-662 (2021)
PMID: 33769681 - Nagashima S, Maruyama J, Honda K, Kondoh Y, Osada H, Nawa M, Nakahama KI, Ishigami-Yuasa M, Kagechika H, Sugimura H, Iwasa H, Arimoto-Matsuzaki K, Nishina H, Hata Y.
CSE1L promotes nuclear accumulation of transcriptional coactivator TAZ and enhances invasiveness of human cancer cells.
J Biol. Chem. 297, 100803 (2021)
PMID: 34022224 - Takayama KI, Honma T, Suzuki T, Kondoh Y, Osada H, Suzuki Y, Yoshida M, Inoue S.
Targeting Epigenetic and Posttranscriptional Gene Regulation by PSF Impairs Hormone Therapy-Refractory Cancer Growth.
Cancer Res. 81, 3495-3508 (2021)
PMID: 33975881 - Yoshida K, Kondoh Y, Nakano T, Bolortuya B, Kawabata S, Iwahashi F, Nagano E, Osada H.
New abscisic acid derivatives revealed adequate regulation of stomatal, transcriptional, and developmental responses to conquer drought.
ACS Chem. Biol. 16, 1566-1575 (2021)
PMID: 34379974 - *Pradipta A R, *Tanaka K.
Biofunctional chemistry and reactivity of biogenic acrolein for cancer diagnosis and therapy.
Chem. Commun. 57, 9798-9806 (2021)
PMID: 34581321 - Nakatani K, Maehama T, Nishio M, Otani J, Yamaguchi K, Fukumoto M, Hikasa H, Hagiwara S, Nishina H, Mak TW, Honma T, Kondoh Y, Osada H, Yoshida M, Suzuki A.
Alantolactone is a natural product that potently inhibits YAP1/TAZ through promotion of reactive oxygen species accumulation.
Cancer Sci. 112, 4303-4316 (2021)
PMID: 34289205 - Sakamoto T, Fukui Y, Kondoh Y, Honda K, Shimizu T, Hara T, Hayashi T, Saitoh Y, Murakami Y, Inoue J, Kaneko S, Osada H, Seiki M.
Pharmacological inhibition of Mint3 attenuates tumour growth, metastasis, and endotoxic shock.
Commun. Biol. 4, 1165 (2021)
PMID: 34621018 - Moon JY, Adams E, Miyazaki T, Kondoh Y, Muroi M, Watanabe N, Osada H, Shin R.
Cesium tolerance is enhanced by a chemical which binds to BETA- GLUCOSIDASE 23 in Arabidopsis thaliana.
Sci. Rep. 11, 21109 (2021)
PMID: 34702872 - Yoshida J, Ohishi T, Abe H, Ohba S, Inoue H, Usami I, Amemiya M, Oriez R, Sakashita C, Dan S, Sugawara M, Kawaguchi T, Ueno J, Asano Y, Ikeda A, Takamatsu M, Amori G, Kondoh Y, Honda K, Osada H, Noda T, Watanabe T, Shimizu T, Shibasaki M, Kawada M.
Mitochondrial complex I inhibitors suppress tumor growth through concomitant acidification of the intra- and extracellular environment.
iScience 24, 103497 (2021)
PMID: 34934919 - Yoshioka H, Kawamura T, Muroi M, Kondoh Y, Honda K, Kawatani M, Aono H, Waldmann H, Watanabe N, Osada H.
Identification of a small molecule that enhances ferroptosis via inhibition of FSP1.
ACS Chem. Biol. 17, 483-491 (2022)
PMID: 35128925
プレスリリース - *Pradipta A R, Michiba H, Kubo A, Fujii M, Tanei T, Morimoto K, Shimazu K, *Tanaka K.
The secon-generation click-to-sense probe for intraoperative diagnosis of breast cancer tissues based on acrolein targeting.
Bull. Chem. Soc. Jpn. (2022) - Chhipi-Shrestha JK, Schneider-Poetsch T, Suzuki T, Mito M, Khan K, Dohmae N, *Iwasaki S, *Yoshida M.
Splicing modulators elicit global translational repression by condensate-prone proteins translated from introns.
Cell Chem. Biol. 29, 259-275 (2022)
PMID: 34520743
プレスリリース - Sasaki K, Suzuki M, Sonoda T, Schneider-Poetsch T, Ito A, Takagi M, Fujishiro S, Sohtome Y, Dodo K, Umehara T, Aburatani H, Shin-ya K, Nakao Y, Sodeoka M, *Yoshida M.
Visualization of the dynamic interaction between nucleosomal histone H3K9 tri-methylation and HP1α chromodomain in living cells.
Cell Chem. Biol. 29, 1153-1161 (2022)
PMID: 35728598
プレスリリース - Kato H, Nemoto K, Shimizu M, Abe A, Asai S, Ishihama N, Matsuoka S, Daimon T, Ojika M, Kawakita K, Onai K, Shirasu K, Yoshida M, Ishiura M, Takemoto D, Takano Y, Terauchi R.
Recognition of pathogen-derived sphingolipids in Arabidopsis.
Science. 376, 857-860 (2022)
PMID: 35587979
プレスリリース - Chhipi-Shrestha JK, Yoshida M, Iwasaki S.
Filter trapping protocol to detect aggregated proteins in human cell lines.
STAR Protoc. 3, 101571 (2022)
PMID: 35880124 - Manzoor M, Muroi M, Ogawa N, Kobayashi H, Nishimura H, Chen D, Fasina OB, Wang J, Osada H, Yoshida M, Xiang L, Qi J.
Isoquercitrin from Apocynum venetum L. produces an anti-obesity effect on obese mice by targeting C-1-tetrahydrofolate synthase, carbonyl reductase, and glutathione S-transferase P and modification of the AMPK/SREBP-1c/FAS/CD36 signaling pathway in mice in vivo.
Food Funct. 13, 10923-10936 (2022)
PMID: 36205648
関連する代表的な論文
- Nishimura S, Arita Y, Honda M, Iwamoto K, Matsuyama A, Shirai A, Kawasaki H, Kakeya H, Kobayashi T, Matsunaga S, *Yoshida M.
Marine antifungal theonellamides targets 3ß-hydroxysterol to activate Rho1 signaling.
Nat. Chem. Biol. 6, 519-526 (2010)
PMID: 20543850 - Hirohama M, Kumar A, Fukuda I, Matsuoka S, Igarashi Y, Saitoh H, Takagi M, Shin-ya K, Honda K, Kondoh Y, Saito T, Nakao Y, Osada H, Zhan KY, Yoshida M, *Ito A.
Spectomycin B1 as a novel SUMOylation inhibitor that directly binds to SUMO E2.
ACS Chem. Biol. 8, 2635-2642 (2013)
PMID: 24143955 - Arita Y, Nishimura S, Ishitsuka R, Kishimoto T, Ikenouchi J, Ishii K, Umeda M, Matsunaga S, Kobayashi T, *Yoshida M.
Targeting cholesterol in a liquid-disordered environment by theonellamides modulates cell membrane order and cell shape.
Chem. Biol. 22, 604-610 (2015)
PMID: 25960262 - Ito A, Shimazu T, Maeda S, Shah AA, Tsunoda T, Iemura S-I, Natsume T, Suzuki T, Motohashi H, Yamamoto M, *Yoshida M.
The subcellular localization and activity of cortactin is regulated by acetylation and interaction with Keap1.
Sci. Signal. 8, ra120 (2015)
PMID: 26602019 - Kondoh Y, Honda K, Hiranuma S, Hayashi T, Shimizu T, Watanabe N, *Osada H.
Comparative chemical array screening for p38γ/δ MAPK inhibitors using a single gatekeeper residue difference between p38α/β and p38γ/δ.
Sci. Rep. 6, 29881 (2016)
PMID: 27431267 - Sun X, Hirai G, Ueki M, Hirota H, Wang Q, Hongo Y, Nakamura T, Hitora Y, Takahashi H, Sodeoka M, Osada H, Hamamoto M, *Yoshida M, *Yashiroda Y.
Identification of novel secreted fatty acids that regulate nitrogen catabolite repression in fission yeast.
Sci. Rep. 6, 20856 (2016)
PMID: 26892493 - Nakaoka S, Sasaki K, Ito A, Nakao Y, *Yoshida M.
A genetically encoded FRET probe to detect intranucleosomal histone H3K9 or H3K14 acetylation using BRD4, a BET family member.
ACS Chem. Biol. 11, 729-733 (2016)
PMID: 25946208 - Takase S, Kurokawa R, Arai D, Kanemoto Kanto K, Okino T, Nakao Y, Kushiro T, *Yoshida M, *Matsumoto K.
A quantitative shRNA screen identifies ATP1A1 as a gene that regulates cytotoxicity by aurilide B.
Sci. Rep. 7, 2002 (2017)
PMID: 28515454 - Yoshimoto R, Kaida D, Furuno M, Burroughs AM, Noma S, Suzuki H, Kawamura Y, Hayashizaki Y, Mayeda A, *Yoshida M.
Global analysis of pre-mRNA subcellular localization following splicing inhibition by spliceostatin A.
RNA 23, 47-57 (2017)
PMID: 27754875 - Piotrowski JS, Li SC, Deshpande R, Simpkins SW, Nelson J, Yashiroda Y, Barber JM, Safizadeh H, Wilson E, Okada H, Gebre AA, Kubo K, Torres N, Leblanc MB, Andrusiak K, Okamoto R, Yoshimura M, van Leeuwen J, Shirahige K, Baryshnikova A, Brown G, Saito T, Costanzo M, Andrews B, Ohya Y, *Osada H, *Yoshida M, *Myers CL, *Boone C.
Functional annotation of chemical libraries across diverse biological processes.
Nat. Chem. Biol. 13, 982-993 (2017)
PMID: 28759014





