林 剛介
ポリユビキチン鎖およびユビキチン化タンパク質の高効率化学合成
 |
研究代表者 林 剛介名古屋大学大学院 工学研究科 生命分子工学専攻 生体分子応用化学研究室 准教授 |
|---|
研究概要
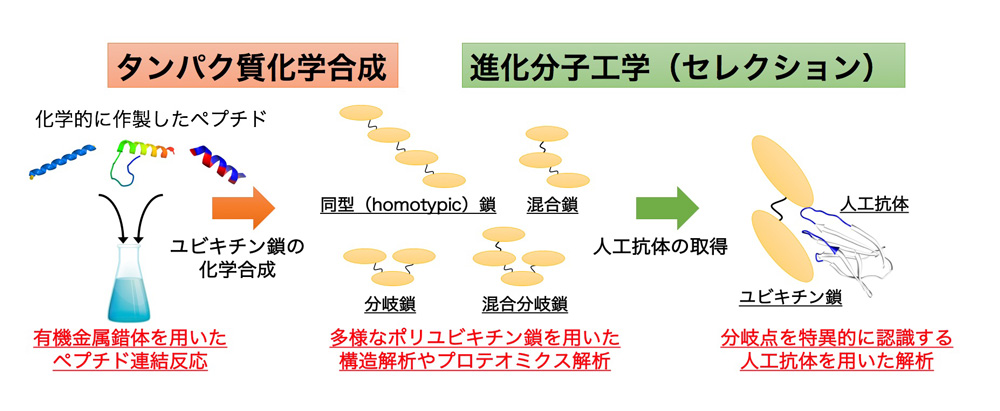
ユビキチンなどのタンパク質はアミノ酸が精密に重合した生体高分子ですが、近年は生体のタンパク質合成システムを利用した遺伝子工学的な手法だけでなく、有機化学を駆使した「タンパク質化学合成法」でも作られるようになってきています。これは、固相担体上でアミノ酸を一つずつ伸長させて数十残基のペプチドを作る「ペプチド固相合成」と、合成したペプチド同士を繋ぎ合わせる「ペプチド連結反応」の発展による結果だと言えます。ペプチド固相合成では、導入可能なアミノ酸の種類がとても多いため、遺伝子工学法では作ることが困難なタンパク質(例えば、翻訳後修飾を持つものや蛍光色素などの機能性分子が導入されたもの)を、化学合成法では作ることができます。これまでに私の研究グループでは、タンパク質化学合成法をより洗練された技術へと昇華させるための分子技術の開発、および化学合成法を用いた修飾タンパク質の作製とその機能解析を行ってきました。その例として、有機金属錯体やDNAを用いた新たなペプチド連結法の開発、また翻訳後修飾や蛍光修飾が施されたヒストンタンパク質の合成とその機能解析などが挙げられます。
本研究では「ユビキチンコード」の解読に寄与するケモテクノロジーとして、既存の方法では作製困難なポリユビキチン鎖およびユビキチン化タンパク質の化学合成法の開発を行います。さらに進化分子工学的手法を融合し、合成したユビキチン鎖やユビキチン化タンパク質に選択的に結合するペプチドあるいは人工抗体の取得を試みます。また、合成したユビキチン鎖やユビキチン化タンパク質を領域内外の研究者に提供し、構造解析やプロテオミクス解析に応用することで、本領域の発展に貢献します。
- 有機金属錯体を用いたポチユビキチン鎖の効率的合成法の開発
化学合成法では、標的とするタンパク質が大きいほどペプチド連結反応の回数が増加し、それに伴って精製回数が増加するため、合成収率および効率の低下が問題となります。そこで本研究では、私たちがこれまでに開発してきた有機金属錯体を用いたone-potペプチド連結法をさらに発展させ、標的とするポリユビキチン鎖およびユビキチン化タンパク質を高効率で合成可能な系の開発を行います。 - ユビキチン鎖の分岐点を認識する人工抗体の取得
化学合成したユビキチン鎖に対して、鎖の分岐パターン特異的に結合する人工抗体(あるいはペプチドアプタマー)の取得を行います。取得した人工抗体は、阻害剤としての利用やプルダウン解析に応用するなどしてユビキチンコードの解読に役立てます。
研究概要を示す模式図
本領域での研究成果
- Yanase M, Nakatsu K, Cardos C J, Konda Y, *Hayashi G, *Okamoto A.
Cysteinylprolyl Imide (CPI) Peptide: A Highly Reactive and Easily Accessible Crypto-thioester for Chemical Protein Synthesis.
Chem. Sci. 10, 5967-5975 (2019)
PMID: 31360403 - Kamo N, *Hayashi G, *Okamoto A.
Chemical Synthesis of Cys-Containing Protein via Chemoselective Deprotection with Different Palladium Complexes.
Org. Lett. 21, 8378-8382 (2019)
PMID: 31560553 - Nakatsu K, Yanase M, *Hayashi G, *Okamoto A.
Fmoc-Compatible and C-terminal-Sequence-Independent Peptide Alkyl Thioester Formation Using Cysteinylprolyl Imide.
Org. Lett. 22, 4670-4674 (2020)
PMID: 32484687 - Kondo T, Iwatani Y, Matsuoka K, Fujino T, Umemoto S, Yokomaku Y, Ishizaki K, Kito S, Sezaki T, Hayashi G, *Murakami H.
Antibody-like proteins that capture and neutralize SARS-CoV-2.
Sci. Adv. 6, eabd3916 (2020)
PMID: 32948512 - Hao F, Murphy K. J, Kujirai T, Kamo N, Kato J, Koyama M, Okamoto A, Hayashi G, Kurumizaka H, Hayes J. J.
Acetylation-modulated communication between the H3 N-terminal tail domain and the intrinsically disordered H1 C-terminal domain.
Nucl. Acids Res. 48, 11510-11520 (2020)
PMID: 33125082 - Kondo T, Eguchi M, Kito S, Fujino T, Hayashi G, Murakami H.
cDNA TRAP display for rapid and stable in vitro selection of antibody-like proteins.
Chem. Commun. 57, 2416-2419 (2021)
PMID: 33554979 - Kamo N, Kujirai T, Kurumizaka H, Murakami H, *Hayashi G, *Okamoto A.
Organoruthenium-Catalyzed Chemical Protein Synthesis to Elucidate the Functions of Epigenetic Modifications on Heterochromatin Factors.
Chem. Sci. AOP (2021)
代表的な論文
- Sueoka T, Hayashi G, *Okamoto A.
Regulation of the Stability of the Histone H2A-H2B Dimer by H2A Tyr57 Phosphorylation.
Biochemistry 56, 4767-4772 (2017)
PMID: 28813589 - Kamo N, Hayashi G, *Okamoto A.
Efficient peptide ligation between allyl-protected Asp and Cys followed by palladium-mediated deprotection.
Chem. Commun. 54, 4337-4340 (2018)
PMID: 29644365 - Sueoka T, Koyama K, Hayashi G, *Okamoto A.
Chemistry-driven Epigenetic Investigation of Histone and DNA Modifications.
Chem. Rec. 18,1727-1744 (2018)
PMID: 30070422 - Kamo N, Hayashi G, *Okamoto A.
Triple Function of 4-Mercaptophenylacetic Acid Promotes One-Pot Multiple Peptide Ligation.
Angew. Chem. Int. Ed. 57, 16533-16537 (2018)
PMID: 30346110 -
Hayashi G, Yanase M, Nakatsuka Y, *Okamoto A.
Simultaneous and Traceless Ligation of Peptide Fragments on DNA Scaffold.
Biomacromolecules 20, 1246-1253 (2019)
PMID: 30677290





