佐藤 裕介
酵素反応と化学反応を駆使した分岐鎖認識の構造生物学
 |
研究代表者 佐藤 裕介鳥取大学大学院 工学研究科 構造生物学研究室 講師 |
|---|
研究概要
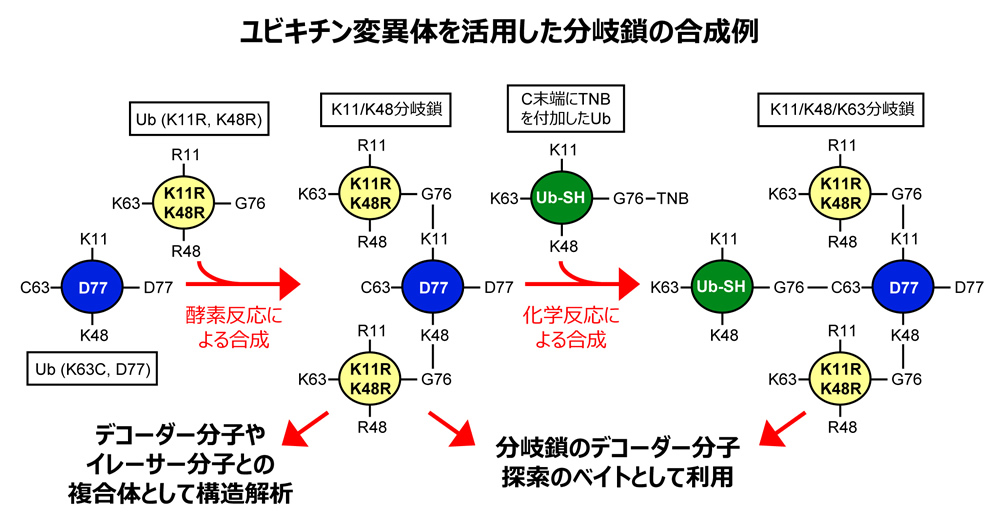
ユビキチン化は翻訳後修飾の一つですが、他の多くの翻訳後修飾因子との大きな違いとしてユビキチン分子が数珠状に次々と連結することでユビキチン鎖が形成されるという点が挙げられます。このユビキチン間の結合は8種類の残基(K6, K11, K27, K29, K33, K48, K68, M1)が使われます。ユビキチン鎖は連結に用いられた残基によって異なる構造を持ち、異なるデコーダー分子に認識されるため、ユビキチンという同一の分子を使用しているにも関わらずユビキチン修飾は生体内において多様な生命現象を制御する事が可能となります。これまでの構造研究により、デコーダー分子によるユビキチン鎖の特異的認識機構は数多く報告されてきました。一方、途中でユビキチン鎖が枝分かれする『分岐鎖』については、細胞内での機能が徐々に報告されはじめ、その重要性が指摘されているにも関わらず、デコーダー分子やイレーサー分子による分岐鎖特異的認識機構は未だ解明されておりません。
本研究では、デコーダー分子やイレーサー分子による分岐鎖特異的認識機構をX線結晶構造解析により解明するため、酵素反応と化学的手法を組み合わせて分岐鎖を大量に合成する方法を確立します。これまで、特定のユビキチン鎖だけを選択的に伸長する酵素は多数発見されているため、これら酵素の組み合わせで分岐鎖を合成することは可能です。しかし、野生型のユビキチンを用いると分岐の位置やユビキチン鎖の長さを制御することは困難です。そこで、望まないユビキチン鎖の伸長を防ぐため、いくつかのリジン残基に変異を導入したユビキチンを使用することで、分岐鎖の制御を行う事ができると考えています。一方、分岐に使用される残基の組み合わせによっては、先に付加されたユビキチンが次のユビキチンの付加を阻害するために、酵素による伸長反応が起こりづらい事が知られています。そのような場合は、ジスルフィド結合を利用した化学的手法によりユビキチンを連結し、擬似的なユビキチン鎖の合成を行う予定です。この方法で合成される分岐鎖は変異を含んだものですが、多くのデコーダー分子やイレーザー分子はリジン残基を認識しない事が知られているため、構造研究を行う上では問題ないと考えています。分岐鎖の大量調製が可能になれば、すでに分岐鎖に対する特異性が報告されているデコーダー分子やイレーサー分子との複合体として構造解析を行う他、分岐鎖のデコーダー分子探索のベイトとしての利用など、様々な利用が可能になることが期待できます。
研究概要を示す模式図
本領域での研究成果
- Li Y, Okatsu K, Fukai S, *Sato Y.
Structural basis for specific recognition of K6-linked polyubiquitin chains by the TAB2 NZF domain.
Biophys J. 120, 3355-3362 (2021)
PMID: 34242591
プレスリリース - *Nakazawa S, Mamiya R, Kawabata-Iwakawa R, Oikawa D, Kaira K, Tokunaga F, Nobusawa S, Sato Y, Sasaki A, Yajima T, Shirabe K.
Identification and molecular analysis of RNF31 Q622H germline polymorphism.
Oncol Lett. 24, 394 (2022)
PMID: 36276481 - Saito R, Tada Y, Oikawa D, Sato Y, Seto M, Satoh A, Kume K, Ueki N, Nakashima M, Hayashi S, Toyoshima Y, Tokunaga F, Kawakami H, *Kakita A.
Spinocerebellar ataxia type 17-digenic TBP/STUB1 disease: neuropathologic features of an autopsied patient.
Acta Neuropathol Commun. 10, 177 (2022)
PMID: 36476347
代表的な論文
- Sato Y, Yoshikawa A, Yamagata A, Mimura H, Yamashita M, Ookata K, Nureki O, Iwai K, Komada M, *Fukai S.
Structural basis for specific cleavage of Lys 63-linked polyubiquitin chains.
Nature 455, 358-362 (2008)
PMID: 18758443 - Sato Y, Fujita H, Yoshikawa A, Yamashita M, Yamagata A, Kaiser SE, *Iwai K, *Fukai S.
Specific recognition of linear ubiquitin chains by the Npl4 zinc finger (NZF) domain of the HOIL-1L subunit of the linear ubiquitin chain assembly complex.
Proc. Natl. Acad. Sci. U.S.A. 108, 20520-20525 (2011)
PMID: 22139374 - Sato Y, Goto E, Shibata Y, Kubota Y, Yamagata A, Goto-Ito S, Kubota K, Inoue J, Takekawa M, Tokunaga F, *Fukai S.
Structures of CYLD USP with Met1- or Lys63-linked diubiquitin reveal mechanisms for dual specificity.
Nat. Struct. Mol. Biol. 22, 222-229 (2015)
PMID: 25686088 - Sato Y, Okatsu K, Saeki Y, Yamano K, Matsuda N, Kaiho A, Yamagata A, Goto-Ito S, Ishikawa M, Hashimoto Y, Tanaka K, *Fukai S.
Structural basis for specific cleavage of Lys6-linked polyubiquitin chains by USP30.
Nat. Struct. Mol. Biol. 24, 911-919 (2017)
PMID: 28945247 - Sato Y, Tsuchiya H, Yamagata A, Okatsu K, Tanaka K, *Saeki Y, *Fukai S.
Structural insights into ubiquitin recognition and Ufd1 interaction of Npl4.
Nat. Commun. 10, 5708 (2019)
PMID: 31836717





