寺井 琢也
進化分子工学によるユビキチン化酵素基質の同定と選択的タンパク質分解法の開発
 |
研究代表者 寺井 琢也東京大学大学院 理学系研究科 化学専攻 生体分子化学研究室 准教授 |
|---|
研究概要
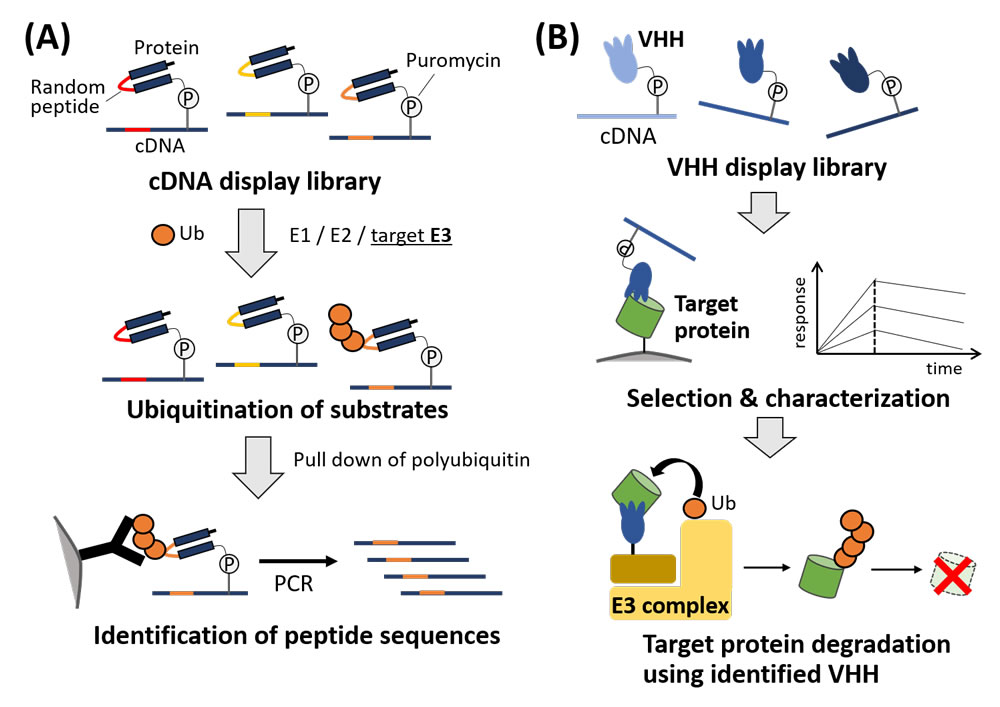
近年、ユビキチンによる細胞内タンパク質の修飾が想像以上に多様性に富むことが明らかになりつつあります。しかしその多様性の源泉である多種多様なユビキチンリガーゼ(E3)の基質特異性に関する知見は未だ不十分です。そこで本研究では、研究代表者が近年取り組んでいるペプチド進化分子工学、特にcDNA display法(=ピューロマイシンDNAリンカーを介してペプチドとそれをコードするcDNAを1分子レベルで共有結合させる技術)を活用して、「特定のE3に選択的に認識・分解されるペプチド配列の網羅的探索」を目指します。具体的にはランダム化したペプチド配列を含むモデルタンパク質を無細胞翻訳系によってcDNA displayへと変換し、標的となるE3を含むユビキチン化酵素系中でインキュベーションします。その後、ユビキチン化されたcDNA display分子をアフィニティー精製してPCRを行い、次世代シークエンサーで配列解析を実施する事で、標的E3がどのペプチドを基質とするのかを網羅的に調べます(図1左)。こうして得られたペプチドの情報は、内在性基質の同定や、条件選択的なタンパク質分解技術の開発に繋がると考えられます。
本研究ではまた、「標的タンパク質に結合する新規VHH(=ラクダ科動物に由来する単一ドメイン抗体)の獲得と、タンパク質分解誘導への応用」にも取り組みます。PROTACやSNIPERなどの人工分子を用いた標的タンパク質のユビキチン化と選択的分解(プロテインノックダウン)は創薬の新たな方法論として注目を集めていますが、標的に対する低分子リガンドが未知の場合には分解誘導が難しいという課題もあります。VHHは一般的な抗体よりサイズが小さく細胞内でも利用できる上、cDNA displayを始めとする進化分子工学やタンパク質工学の対象としても適しています。本研究ではまず、VHHの大規模ライブラリーから試験管内淘汰によって細胞内標的タンパク質に結合する分子を探索します。こうして得られた人工抗体を認識部位として用いれば、任意のタンパク質に対するノックダウンができるはずです(図1右)。さらに、VHHに光応答性や低分子応答性を付与する研究と組み合わせることで、最終的には刺激に応答して可逆的にタンパク質分解を制御することも可能になると期待されます。
研究概要を示す模式図
本領域での研究成果
- Anzai H, *Terai T, Wakabayashi-Nakao K, Noguchi T, Kumachi S, Tsuchiya M, *Nemoto N.
Interleukin-17A Peptide Aptamers with an Unexpected Binding Moiety Selected by cDNA Display under Heterogenous Conditions.
ACS Med. Chem. Lett. 12, 1427-1434 (2021)
PMID: 34531951 - Zhu W, Takeuchi S, Imai S, Terada T, Ueda T, Nasu Y, *Terai T, *Campbell R E.
Chemigenetic indicators based on synthetic chelators and green fluorescent protein.
Nat. Chem. Biol. 19, 38-44 (2023)
PMID: 36138142
プレスリリース
代表的な論文
- 寺井琢也、熊地重文、*根本直人: 「cDNA display法によるペプチドスクリーニング技術」ペプチド医薬品のスクリーニング・安定化・製剤化技術(技術情報協会編)40-49 (2017)
- Kobayashi S, Terai T, Yoshikawa Y, Ohkawa R, Ebihara M, Hayashi M, Takiguchi K, *Nemoto N.
In vitro selection of random peptides against artificial lipid bilayers: a potential tool to immobilize molecules on membranes.
Chem. Commun.53, 3458–3461 (2017)
PMID: 28271115 - *Terai T, Anzai H, *Nemoto N.
Selection of Peptides that Associate with Dye-Conjugated Solid Surfaces in a pH-Dependent Manner Using cDNA Display.
ACS Omega4, 7378-7384 (2019)
PMID: 31459836 - Jayathilake C, Kumachi S, Arai H, Motohashi M, Terai T, Murakami A, *Nemoto N.
In vitro selection of anti-gliadin single-domain antibodies from a naïve library for cDNA-display mediated immuno-PCR.
Anal. Biochem. 589, 113490 (2020)
PMID: 31678363 - *Terai T, Koike T, *Nemoto N.
Photocrosslinking of cDNA Display Molecules with Their Target Proteins as a New Strategy for Peptide Selection.
Molecules25, 1472 (2020)
PMID: 32214008





