研究チーム
Team研究チーム
研究項目A01 計画研究:ケモテクノロジーによるユビキチンコードの解読と制御
ケモテクノロジーと質量分析計を活用したユビキチンコードの解読
研究代表者 佐伯 泰公益財団法人東京都医学総合研究所 基礎医科学研究分野 蛋白質代謝プロジェクト プロジェクトリーダー |
|
研究分担者 大竹 史明星薬科大学先端生命科学研究所 特任准教授 |
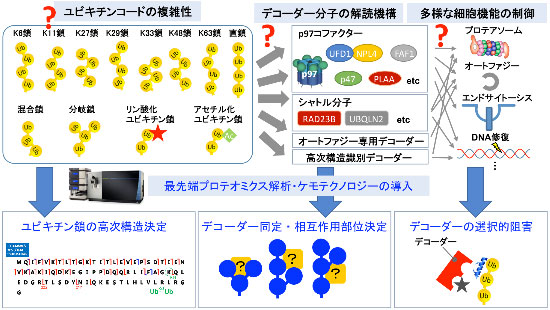
本計画研究の概要
私たちはユビキチン鎖の連結様式に着目した高感度定量プロテオミクス解析を開発し、様々な鎖の機能を解析してきました。その過程でリン酸化やアセチル化などの化学修飾を受けたユビキチンを同定するなどユビキチンコードの拡大に貢献するとともに、ユビキチン鎖の「長さ」や「分岐」といった高次構造がユビキチンコードの機能において極めて重要な役割を果たす可能性を世界に先駆けて提唱してきました。しかし、ユビキチンコードの全体像は未だ不明であり、デコーダー分子の使い分けや機能連携も不明な点が数多く残されています。
本研究では、ユビキチン解析に特化した様々な最先端プロテオミクス解析法を確立し、領域内で開発するケモテクノロジーを利用して、ユビキチンシグナルの根幹である「ユビキチン修飾の高次構造」の直接の解析と「デコーダー分子による識別・解読機構」の解明に挑戦します。
1. 最先端ユビキチン・プロテオミクス解析法の開発
最新鋭の超高感度高性能質量分析計を用いて世界最先端のプロテオミクス解析法をユビキチン研究に導入します。具体的には、ユビキチン化基質の網羅的変動解析、架橋剤を用いたタンパク質複合体相互作用部位決定、ユビキチン鎖のトップダウン解析、ユビキチン鎖の超高感度絶対定量解析などの研究手法を開発します。そして、新たなユビキチンコード、デコーダー分子を探索するとともに、領域内の全研究を支援します。
2. ユビキチンデコーダー分子のユビキチン鎖タイプ選択性の解析
さまざまなユビキチン依存的経路を制御するAAA型シャペロンp97のユビキチン結合コファクターを中心に、ユビキチン鎖タイプ選択性と基質タンパク質について解明します。
3. ケモテクノロジーを利用したユビキチンコードの解析
ユビキチン鎖の機能を喪失させるケミカルツールとユビキチン化基質の網羅的変動解析により、個々のユビキチン鎖の細胞機能を解明します。
4. ケモテクノロジーによるユビキチンデコーダー分子の解析
化学合成ユビキチン鎖を用いて、ユビキチン修飾の高次構造(鎖長や分岐)を識別する新規デコーダー分子を同定し、機能解析を進めます。一方、ユビキチン鎖との相互作用を破壊する阻害ペプチドや化合物を用いて、p97コファクターやプロテアソームのシャトル分子などのデコーダー分子の基質選択性や機能連携を解明します。
研究概要を示す模式図
本領域での研究成果
- Tomita T, Hirayama S, Sakurai Y, Ohte Y, Yoshihara H, Saeki Y, Hamazaki J, *Murata S.
Specific modification of aged proteasomes revealed by tag-exchangeable knock-in mice.
Mol. Cell. Biol. 39, e00426-18 (2018)
PMID: 30348842 - Ikeuchi K, Tesina P, Matsuo Y, Sugiyama T, Cheng J, Saeki Y, Tanaka K, Becker T, *Beckmann R, *Inada T.
Collided ribosomes form a unique structural interface to induce Hel2-driven quality control pathways.
EMBO J. 38, e100276 (2019)
PMID: 30609991 - *Masuda Y, Saeki Y, Arai N, Kawai H, Kukimoto I, Tanaka K, *Masutani C.
Stepwise multipolyubiquitantion of p53 by the E6AP-E6 ubiquitin ligase complex.
J. Biol. Chem. 294, 14860-14875 (2019)
PMID: 31492752 - Sato Y, Tsuchiya H, Yamagata A, Okatsu K, Tanaka K, *Saeki Y, *Fukai S.
Structural insights into ubiquitin recognition and Ufd1 interaction of Npl4.
Nat. Commun. 10, 5708 (2019)
PMID: 31836717
プレスリリース
http://www.iam.u-tokyo.ac.jp/press_release/191213/
http://www.igakuken.or.jp/topics/2019/1213.html
http://www.igakuken.or.jp/english/topics/2019/e-1213.html - Yasuda S, Tsuchiya H, Kaiho A, Guo Q, Ikeuchi K, Endo A, Arai N, Ohtake F, Murata S, Inada T, Baumeister W, Fernandez-Busnadiego R, *Tanaka K, *Saeki Y.
Stress- and ubiquitylation-dependent phase separation of the proteasome.
Nature 578, 296-300 (2020)
PMID: 32025036
プレスリリース
http://www.igakuken.or.jp/topics/2020/0205.html (都医学研)
https://www.amed.go.jp/news/release_20200206-02.html(AMED)
Highlighted in Cell Res
EurekAlert!
Faculty Opinions - *Nishiyama A, Mulholland C, Bultmann S, Kori A, Endo A, Saeki Y, Qin W, Trummer C, Chiba Y, Yokoyama H, Kumamoto S, Kawakami T, Hojo H, Nagae G, Aburatani H, Tanaka K, *Arita K, *Leonhardt H, *Nakanishi M.
Two distinct modes of DNMT1 recruitment ensure the stable maintenance DNA methylation.
Nat. Commun. 11, 1222 (2020)
PMID: 32144273
プレスリリース
Faculty Opinions - Matsuo Y, Tesina P, Nakajima S, Mizuno M, Endo A, Buschauer R, Cheng J, Shounai O, Ikeuchi K, Iwasaki S, Saeki Y, Becker T, *Beckmann R, *Inada T.
RQT complex dissociates ribosomes collided on endogenous RQC substrate SDD1.
Nat. Stuct. Mol. Biol. 27, 323-332 (2020)
PMID: 32203490 - Oikawa D, Sato Y, Ohtake F, Komakura K, Hanada K, Sugawara K, Terawaki S, Mizukami Y, Phuong HT, Iio K, Obika S, Fukushi M, Irie T, Tsuruta D, Sakamoto S, Tanaka K, Saeki Y, Fukai S, *Tokunaga F.
Molecular bases for HOIPINs-mediated inhibition of LUBAC and innate immune responses.
Commun. Biol. 3, 163 (2020)
PMID: 32246052 - Fuseya Y, Fujita H, Kim M, Ohtake F, Nishide A, Sasaki K, Saeki Y, Tanaka K, Takahashi R, *Iwai K.
The HOIL-1L ligase modulates immune signalling and cell death via monoubiquitination of LUBAC.
Nat. Cell Biol. 22, 663-673 (2020)
PMID: 32393887 - *Suzuki G, Imura S, Hosokawa M, Katsumata R, Nonaka T, Hisanaga SI, Saeki Y, *Hasegawa M.
α-Synuclein strains that cause distinct pathologies differentially inhibit proteasome.
eLife 9, e56825 (2020)
PMID: 32697196 - *Watanabe M, Saeki Y, Takahashi H, Ohtake F, Yoshida Y, Kasuga Y, Kondo T, Yaguchi H, Suzuki M, Ishida H, Tanaka K, *Hatakeyama S.
A substrate-trapping strategy to find E3 ubiquitin ligase substrates identifies Parkin and TRIM28 targets.
Commun. Biol. 3,592 (2020)
PMID: 33082525 - Matsuki Y, Matsuo Y, Nakano Y, Iwasaki S, Yoko H, Udagawa T, Li S, Saeki Y, Yoshihisa T, Tanaka K, Ingolia N, *Inada T.
Ribosomal protein S7 ubiquitination during ER stress in yeast is associated with selective mRNA translation and stress outcome.
Sci. Rep. 10, 19669 (2020)
PMID: 33184379 - Nakabayashi O, Takahashi H, Moriwaki K, Komazawa-Sakon S, Ohtake F, Murai Y, Koyahara Y, Saeki Y, Yoshida Y, Yamazaki S, Tokunaga F, Sawasaki T, *Nakano H.
MIND bomb 2 prevents RIPK1 kinase activity-dependent and -independent apoptosis through ubiquitylation of cFLIPL.
Commun. Biol. 4, 80 (2021)
PMID: 33469115 - Kaiho-Soma A, Akizuki Y, Igarachi K, Endo A, Shoda T, Kawase Y, Demizu Y, Naito M, Saeki Y, Tanaka K, *Ohtake F.
TRIP12 promotes small molecule-induced degradation of BRD4 through K29/K48 branched ubiquitin chains.
Mol. Cell 81, 1411-1424.e7 (2021)
PMID: 33567268
プレスリリース - Imada T, Shimi T, Kaio A, Saeki Y, *Kimura H.
RNA polymerase II condensate formation and association with Cajal and histone locus bodies in living human cells.
Genes Cells 26, 298-312 (2021)
PMID: 33608942 - Yokoo H, *Shibata N, Endo A, Ito T, Yanase Y, Murakami Y, Fujii K, Hamamura K, Saeki Y, *Naito M, *Aritake K, *Demizu Y.
Discovery of a highly potent and selective PROTAC targeting hematopoietic prostaglandin D synthase via in silico design.
J. Med. Chem. 64, 15868-15882 (2021)
PMID: 34652145 - Huang Y, Yokoe H, Kaiho-Soma A, Takahashi K, Hirasawa Y, Morita H, *Ohtake F, *Kanoh N.
Design, Synthesis, and Evaluation of Trivalent PROTACs Having a Functionalization Site with Controlled Orientation.
Bioconjug Chem. 33, 142-151 (2022)
PMID: 34878263 - Tsunoda T, Riku M, Yamada N, Tsuchiya H, Tomita T, Suzuki M, Kizuki M, Inoko A, Ito H, Murotani K, Murakami H, Saeki Y, *Kasai K.
ENTREP/FAM189A2 downregulated in breast cancer encodes a new activator for ITCH ubiquitin ligase.
EMBO Rep. 23, e51182 (2022)
PMID: 34927784 - Hasegawa Y, Reyes TH, Uemura T, Baral A, Fujimaki A, Luo Y, Morita Y, Saeki Y, Maekawa S, Yasuda S, Mukuta K, Fukao Y, Tanaka K, Nakano A, Takagi J, Bhalerao RP, Yamaguchi J, Sato T.
The TGN/EE SNARE protein SYP61 and the ubiquitin ligase ATL31 cooperatively regulate plant responses to carbon/nitrogen conditions in Arabidopsis.
Plant Cell. 34, 1354-1374 (2022)
PMID: 35089338 - Akizuki Y, Morita M, Mori Y, Kaiho-Soma A, Dixit S, Endo A, Shimogawa M, Hayashi G, Naito M, Okamoto A, Tanaka K, Saeki Y, *Ohtake F.
cIAP1-based degraders induce degradation via branched ubiquitin architectures.
Nat Chem Biol. 19, 311-322 (2022)
PMID: 36316570 - Ishigaki H, Yasui F, Nakayama M, Endo A, Yamamoto N, Yamaji K, Nguyen C, Kitagawa Y, Sanada T, Honda T, Munakata T, Higa M, Toyama S, Kono R, Takagi A, Matsumoto Y, Koseki A, Hayashi K, Shiohara M, Ishii K, Saeki Y, *Itoh Y, *Kohara M.
An attenuated vaccinia vaccine encoding the severe acute respiratory syndrome coronavirus-2 spike protein elicits broad and durable immune responses, and protects cynomolgus macaques and human angiotensin-converting enzyme 2 transgenic mice from severe acute respiratory syndrome coronavirus-2 and its variants.
Front. Microbiol. 13, 967019 (2022)
PMID: 36466631
関連する代表的な論文
- Pack CG, Yukii H, Toh-e A, Kudo T, Tsuchiya H, Kaiho A, Sakata E, Murata S, Sako Y, Baumeister W, Tanaka K, *Saeki Y.
Quantitave live-cell imaging reveals spatio-temporal dynamics and cytoplasmic assembly of the 26S proteasome.
Nat. Commun. 5, 3396 (2014)
PMID: 24598877 - Koyano F, Okatsu K, Kosako H, Tamura Y, Go E, Kimura M, Kimura Y, Tsuchiya H, Yoshihara H, Hirokawa T, Endo T, Fon EA, Trempe J, Saeki Y, *Tanaka K,*Matsuda N.
Ubiquitin is phosphorylated by PINK1 to activate parkin.
Nature 510, 162-166 (2014)
PMID: 24784582 - *Ohtake F, Saeki Y, Sakamoto K, Ohtake K, Nishikawa H, Tsuchiya H, Ohta T, Tanaka K, Kanno J.
Ubiquitin acetylation inhibits polyubiquitin chain elongation.
EMBO Rep. 16, 192-201 (2015)
PMID: 25527407 - *Yoshida Y, Saeki Y, Murakami A, Kawawaki J, Tsuchiya H, Yoshihara H, Shindo M, *Tanaka K.
A comprehensive method for detecting ubiquitinated substrates using TR-TUBE.
Proc. Natl. Acad. Sci. U.S.A. 112, 4630-4635 (2015)
PMID: 25827227 - *Ohtake F, Saeki Y, Ishido S, Kanno J, *Tanaka K.
The K48-K63 branched ubiquitin chain regulates NF-kB signaling.
Mol. Cell. 64, 251-266 (2016)
PMID: 27746020 - Matsuo Y, Ikeuchi K, Saeki Y, Iwasaki S, Schmidt C, Udagawa T, Sato F, Tsuchiya H, Becker T, Tanaka K, Ingolia NT, Beckmann R, *Inada T.
Ubiquitination of stalled ribosome triggers ribosome-associated quality control.
Nat. Commun. 8, 159 (2017)
PMID: 28757607 - Sato Y, Okatsu K, Saeki Y, Yamano K, Matsuda N, Kaiho A, Yamagata A, Goto-Ito S, Ishikawa M, Hashimoto Y, Tanaka K, *Fukai S.
Structural basis for specific cleavage of Lys6-linked polyubiquitin chains by USP30.
Nat. Struct. Mol. Biol. 24, 911-919 (2017)
PMID: 28945247 - Tsuchiya H, Ohtake F, Arai N, Kaiho A, Yasuda S, *Tanaka K, *Saeki Y.
In vivo ubiquitin linkage-type analysis reveals that the Cdc48-Rad23/Dsk2 axis contributes to K48-linked chain specificity of the proteasome.
Mol. Cell. 66, 488-502 (2017)
PMID: 28525741 - *Ohtake F, Tsuchiya H, Saeki Y, *Tanaka K.
K63 ubiquitylation triggers proteasomal degradation by seeding branched ubiquitin chains.
Proc. Natl. Acad. Sci. U.S.A. 115, E1401-E1408 (2018)
PMID: 29378950 - Tsuchiya H, Burana D, Ohtake F, Arai N, Kaiho A, Komada M, *Tanaka K, *Saeki Y.
Ub-ProT reveals global length and composition of protein ubiquitylation in cells.
Nat. Commun. 9, 524 (2018)
PMID: 29410401





