大竹 史明
Teamユビキチンコードの修飾間クロストークと細胞応答
研究分担者 大竹 史明星薬科大学先端生命科学研究所 特任准教授 |
研究概要
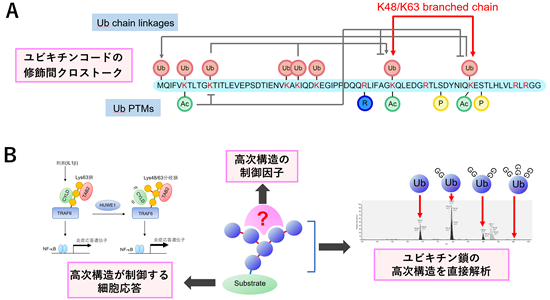
ユビキチンは多様な様式でタンパク質を修飾することで、プロテアソーム依存性のタンパク質分解だけではなく、シグナル伝達やDNA修復、選択的オートファジーなど様々な細胞機能を制御します。ユビキチンは鎖状に連なることで8種類の異なるユビキチン鎖を形成し、それぞれが異なる機能を発現します。このようなユビキチン鎖の多様性に内包された機能情報は「ユビキチンコード」と称されるに至っています。
私達はこれまでに、ユビキチンのアセチル化修飾やK48/K63分岐型ユビキチン鎖を発見し、これら新しいユビキチンコードの機能を解明してきました。K48/K63分岐型ユビキチン鎖を形成するユビキチンリガーゼとしてHUWE1、UBR5を同定し、分岐鎖が炎症シグナル伝達やプロテアソーム依存性分解を制御することを報告しました。これらの研究から、ユビキチン自身の翻訳後修飾やユビキチン鎖の枝分かれなどの高次構造が、ユビキチンコードの「修飾間クロストーク」を生み出していることが明らかとなりました(図A)。しかしながら、複雑なユビキチン修飾の高次構造の解析は技術的な障壁が高く、その機能発現機構については不明な点が数多く残されています。
そこで本分担研究では、質量分析計を用いた定量プロテオミクス技術を駆使することで、ユビキチンコードの機能解明に取り組みます(図B)。ユビキチン鎖の「分岐」や「長さ」といった高次構造を測定する技術開発を試みるととともに、このような高次構造を制御する因子を同定し、ユビキチン鎖の高次構造によって制御される細胞応答を明らかにしていきます。さらに、化合物によるユビキチンリガーゼのハイジャックを利用して、細胞内で分岐鎖を形成させる技術にも取り組んでいきます。また、ユビキチン鎖の絶対定量解析やユビキチン化基質の網羅解析などのユビキチンに特化したプロテオミクス解析法を開発し、領域内の共同研究を推進します。
研究概要を示す模式図
本領域での研究成果
- Ohtake F, Tsuchiya H, Tanaka K, *Saeki Y.
Methods to measure ubiquitin chain length and linkage.
Meth. Enzymol. 618,105-133 (2019)
PMID: 30850048 - Yasuda S, Tsuchiya H, Kaiho A, Guo Q, Ikeuchi K, Endo A, Arai N, Ohtake F, Murata S, Inada T, Baumeister W, Fernandez-Busnadiego R, *Tanaka K, *Saeki Y.
Stress- and ubiquitylation-dependent phase separation of the proteasome.
Nature 578, 296-300 (2020)
PMID: 32025036
プレスリリース
http://www.igakuken.or.jp/topics/2020/0205.html(都医学研)
https://www.amed.go.jp/news/release_20200206-02.html(AMED)
Highlighted in Cell Res
EurekAlert!
Faculty Opinions - Oikawa D, Sato Y, Ohtake F, Komakura K, Hanada K, Sugawara K, Terawaki S, Mizukami Y, Phuong HT, Iio K, Obika S, Fukushi M, Irie T, Tsuruta D, Sakamoto S, Tanaka K, Saeki Y, Fukai S, *Tokunaga F.
Molecular bases for HOIPINs-mediated inhibition of LUBAC and innate immune responses.
Commun. Biol. 3, 163 (2020)
PMID: 32246052 - Fuseya Y, Fujita H, Kim M, Ohtake F, Nishide A, Sasaki K, Saeki Y, Tanaka K, Takahashi R, *Iwai K.
The HOIL-1L ligase modulates immune signalling and cell death via monoubiquitination of LUBAC.
Nat. Cell Biol. 22, 663-673 (2020)
PMID: 32393887 - *Watanabe M, Saeki Y, Takahashi H, Ohtake F, Yoshida Y, Kasuga Y, Kondo T, Yaguchi H, Suzuki M, Ishida H, Tanaka K, *Hatakeyama S.
A substrate-trapping strategy to find E3 ubiquitin ligase substrates identifies Parkin and TRIM28 targets.
Commun. Biol. 3,592 (2020)
PMID: 33082525 - Nakabayashi O, Takahashi H, Moriwaki K, Komazawa-Sakon S, Ohtake F, Murai Y, Koyahara Y, Saeki Y, Yoshida Y, Yamazaki S, Tokunaga F, Sawasaki T, *Nakano H.
MIND bomb 2 prevents RIPK1 kinase activity-dependent and -independent apoptosis through ubiquitylation of cFLIPL.
Commun. Biol. 4, 80 (2021)
PMID: 33469115 - Kaiho-Soma A, Akizuki Y, Igarachi K, Endo A, Shoda T, Kawase Y, Demizu Y, Naito M, Saeki Y, Tanaka K, *Ohtake F.
TRIP12 promotes small molecule-induced degradation of BRD4 through K29/K48 branched ubiquitin chains.
Mol. Cell 81, 1411-1424.e7 (2021)
PMID: 33567268
プレスリリース - Huang Y, Yokoe H, Kaiho-Soma A, Takahashi K, Hirasawa Y, Morita H, *Ohtake F, *Kanoh N.
Design, Synthesis, and Evaluation of Trivalent PROTACs Having a Functionalization Site with Controlled Orientation.
Bioconjug Chem. 33, 142-151 (2022)
PMID: 34878263 - Akizuki Y, Morita M, Mori Y, Kaiho-Soma A, Dixit S, Endo A, Shimogawa M, Hayashi G, Naito M, Okamoto A, Tanaka K, Saeki Y, *Ohtake F.
cIAP1-based degraders induce degradation via branched ubiquitin architectures.
Nat Chem Biol. 19, 311-322 (2022)
PMID: 36316570
代表的な論文
- *Ohtake F, Saeki Y, Sakamoto K, Ohtake K, Nishikawa H, Tsuchiya H, Ohta T, Tanaka K, Kanno J.
Ubiquitin acetylation inhibits polyubiquitin chain elongation.
EMBO Rep. 16, 192-201 (2015)
PMID: 25527407 - *Ohtake F, Saeki Y, Ishido S, Kanno J, *Tanaka K.
The K48-K63 branched ubiquitin chain regulates NF-kB signaling.
Mol. Cell. 64, 251-266 (2016)
PMID: 27746020 - Tsuchiya H, Ohtake F, Arai N, Kaiho A, Yasuda S, *Tanaka K, *Saeki Y.
In vivo ubiquitin linkage-type analysis reveals that the Cdc48-Rad23/Dsk2 axis contributes to K48-linked chain specificity of the proteasome.
Mol. Cell. 66, 488-502 (2017)
PMID: 28525741 - *Ohtake F, Tsuchiya H, Saeki Y, *Tanaka K.
K63 ubiquitylation triggers proteasomal degradation by seeding branched ubiquitin chains.
Proc. Natl. Acad. Sci. U.S.A. 115, E1401-E1408 (2018)
PMID: 29378950





